Bioinformatics Bootcamp — это четырехдневный интенсив, в течение которого участники изучали технологии и инструменты анализа данных с помощью наставников, посещали воркшопы от экспертов и работали над проектами в области геномики и метагеномики. Участники искали новые виды бактерий в экосистеме России, изучали влияние микроорганизмов на здоровье человека, разрабатывали программы для обнаружения вирусов, оценивали влияние мутаций на структуру генов и решали другие задачи.
В хакатоне участвовали студенты, выпускники и аспиранты вузов. Всего приняли участие 120 человек в составе 24 команд из Петербурга, Москвы, Казани, Воронежа, Минска, Ростова-на-Дону и других городов.
Партнерами хакатона выступили компания Genotek, центр ветеринарной генетики Zoogen и центр геномных технологий «Сербалаб».
Все участники хакатона, которые представили свои проекты, смогут без экзаменов поступить в магистратуру «Прикладная геномика» на бюджет после собеседования с руководителем программы, а также в дальнейшем поработать над реальными проектами от лаборатории «Прикладная геномика» и академических и индустриальных партнеров.
Финальный питч проектов состоялся 27 июня — по его итогам жюри выбрало три команды-победителя. Проекты оценивали по четырем критериям: дизайн исследования, результаты исследования, качество презентации и потенциальная польза.
Финальный питч проектов хакатона Bioinformatics Bootcamp. Фото: Дмитрий Григорьев / ITMO.NEWS
Какие решения представили
Второе место с одинаковыми баллами разделили две команды — «Кроны» и My Little Protein.
Исследование состава микробиоты кишечника. Команда «Кроны» исследовала данные образцов толстого кишечника — зоны, которая больше всего насыщена бактериями в организме. Образцы были взяты как у здоровых людей, так и у пациентов с заболеваниями, связанными с воспалением желудочно-кишечного тракта: синдромом раздраженного кишечника, болезнью Крона, язвенным колитом, а также колоректальным раком. Задача состояла в том, чтобы сравнить образцы микробиоты и найти общие и уникальные для разных болезней бактерии в микробиоте пациентов.
Исследователи обнаружили, что у здоровых людей и пациентов с воспалительными заболеваниями кишечника совпадают 114 таксономических уровней бактерий. При этом для каждого заболевания нашлись порядка 3–7% уникальных таксономических уровней. В перспективе результаты работы можно использовать для селективной диагностики заболевания, а также лечения с помощью таргетного воздействия на конкретные бактерии в виде особой диеты, лекарств или фекальной трансплантации, подчеркнула руководительница команды Валерия Косс.
Поиск гена регенерации. Команда My Little Protein исследовала геном иглистой мыши — животного, которое известно своей редкой для млекопитающих способностью к регенерации. Например, мышь может отбросить лоскут кожи или хвост и «отрастить» новые без образования рубцов.
В геноме мыши исследователи искали гены, кодирующие белки, которые могут быть связаны с повышенными регенеративными способностями животного. В результате авторы нашли несколько белков, которые проявляют повышенную экспрессию в ходе заживления повреждений у мышей. Это противовоспалительный белок интерлейкин 10 (Il10) и матрикс-ремоделирующие белки Mmp2 и Mmp9.
«Было бы интересно сравнить наши данные о белках с белками других организмов, которые показывают повышенные способности к регенерации. В дальнейшем эти результаты можно использовать при создании препаратов для заживления», — рассказала участница команды Татьяна Сизова.
Участница хакатона и Татьяна Сизова (слева). Фото: Дмитрий Григорьев / ITMO.NEWS
Создание модели машинного обучения для диагностики гастрита. Первое место заняла команда «Slavные парни». Ребята исследовали наборы данных об образцах кишечной микробиоты пациентов с гастритом из четырех регионов России: центрального, южного, юго-западного и Сибири. Данные включали содержание различных бактерий в образцах микробиоты толстого кишечника. Команда искала взаимосвязь между составом микробиоты людей и регионами их проживания, а также строила модель машинного обучения, которая по анализам микробиоты кишечника будет диагностировать гастрит.
Результаты показали, что место жительства никак не влияет на микробиоту кишечника человека. Средняя точность построенной модели Gastritis.ai составила 97%. С ее помощью исследователи выяснили, какие бактерии могут стать индикаторами гастрита: Faecalibacterium prausnitzii, Bacteroides uniformis и Ruminococcus bromii.
Средняя точность построенной модели Gastritis.ai. Фото предоставлено организаторами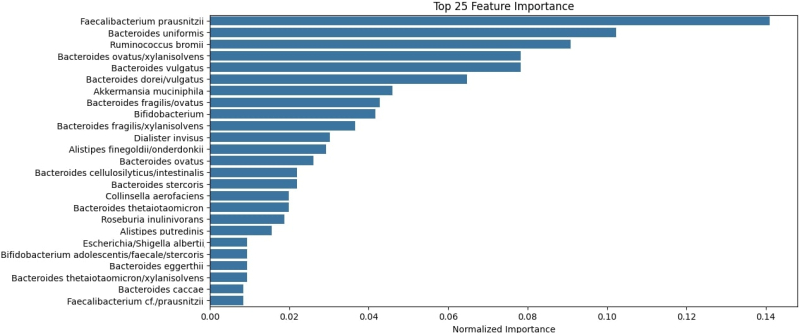
«Кроме модели, которая может помочь с постановкой диагноза, мы смогли вычислить возможные "индикаторы" гастрита. Обычно гастрит вызывает конкретная бактерия — Helicobacter pylori. Мы же смогли идентифицировать болезнь по анализам кишечной микробиоты, в которых этой бактерии нет, выделив бактерии, на которые стоит обращать внимание при постановке диагноза. Изменение их количества может означать развитие заболевания. В перспективе наши результаты можно использовать при диагностике гастрита», — рассказал руководитель команды Евгений Бриленков.




