Миллионы людей по всему миру страдают от разнообразных воспалительных заболеваний кишечника (ВЗК). Это тяжелые хронические болезни, приносящие пациентам периодическую боль, диарею, потерю веса и даже лихорадку. Наиболее распространенными ВЗК сегодня являются язвенный колит (ЯК) и болезнь Крона (БК). По статистике, ЯК страдает каждый двухсотый житель Норвегии, а БК диагностируют практически у каждого трехсотого жителя Германии.
Клиническая картина воспалительных заболеваний кишечника бывает сходна, поэтому очень важно, чтобы в арсенале врачей были надежные способы диагностики, позволяющие определить, чем именно болен человек, и какое лечение ему требуется. Одним из перспективных методов является метагеномный анализ микробиоты кишечника, который позволяет судить о том, какие полезные и вредные микроорганизмы живут в пищеварительном тракте пациента.
Исследованию потенциала этого метода были посвящены международные соревнования Metagenomics diagnosis for infammatory bowel disease challenge (MEDIC) для ученых, занимающихся биоинформатикой, прошедшие в 2019-2020 годах.
Команда, в которую вошли студент Университета ИТМО Артем Иванов и его научный руководитель, возглавляющий Международную лабораторию «Компьютерные технологии», Владимир Ульянцев, смогла занять первое место сразу в двух подсоревнованиях турнира.

От США до Японии
Соревнования проходили в онлайн-формате — чтобы принять в них участие, надо было зарегистрироваться на сайте организаторов до середины января 2020 года.
«Прошлой осенью Владимир Ульянцев показал мне письмо с предложением участвовать в соревновании, — вспоминает Артем Иванов. ― Мне показалось, что это интересно — соревнования, направленные на поиск более точной диагностики для заболеваний. В нашей лаборатории мы уже разрабатывали некоторые программы для метагеномного анализа микробиоты, поэтому мы решили заявиться».
При этом сам Владимир Ульянцев признается, что на момент подачи не верил, что удастся выиграть. Это не удивительно — конкуренция была достаточно высокой: соревнования собрали 26 команд со всего мира. Десять — представляли Италию, по шесть заявилось от Греции и Японии, по три ― от Финляндии и США.

Два в одном
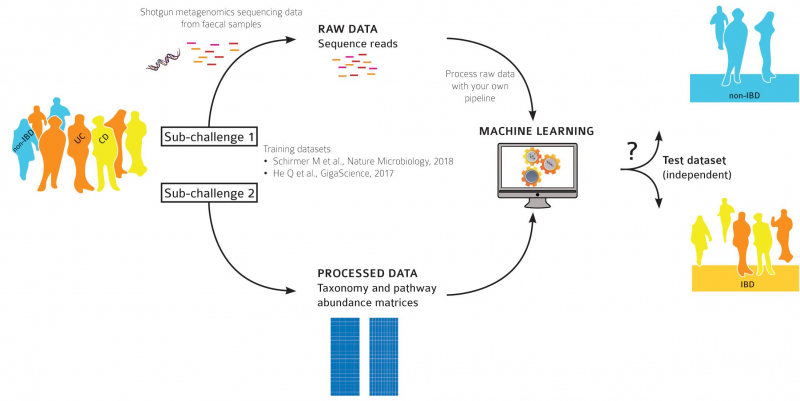
Участники турнира должны были решить две задачи, по каждой из которых велся отдельный зачет. Фактически каждое задание превращалось в отдельное подсоревнование со своими целями, подсчетом очков и победителями.
Оба подсоревнования проходили в стандартном формате для турниров по машинному обучению — сначала участникам давались размеченные данные микробиотного исследования 150 пациентов. На них нужно было обучить компьютерный алгоритм правильно определять состояние человека — здоров ли он, болен ли язвенным колитом или страдает от болезни Крона. После этого обученный анализатор должен был проверить набор таких же данных, только без отметок, и сам сделать вывод, к какой из трех категорий относится каждый пациент.
«В первом соревновании нам было известно, какие бактерии населяют кишечник и в каком количестве, — рассказывает Артем Иванов. — Эти данные не надо было никак предобрабатывать, мы сразу запускали алгоритмы машинного обучения. Мы попробовали разные алгоритмы бустинга, в итоге один из них и сработал. Второе задание было гораздо интереснее — нам давались лишь сырые данные прочтения метагенома — короткие последовательности ДНК микробиоты пациентов, про которые ничего не было известно. Нам самим предстояло выделить признаки, которые являются ключевыми для постановки диагноза, и на их основе сделать предсказание на неразмеченных данных. Мы использовали нашу программу MetaFast, которая из всей информации о ДНК выделяет некоторые связанные структуры, которые и могут являться ключевыми. На основе этих выделенных элементов мы запускали машинное обучение и создавали наш классификатор».
Игра вслепую
Были на MEDIC и отличия от стандартных соревнований по машинному обучению. Как правило, на таких турнирах команды формулируют гипотезу, обучают на ее основе алгоритм, запускают его на неразмеченных данных, отправляют организаторам и в режиме реального времени видят, насколько алгоритм успешно справился с задачей и какое место команды занимают в турнирной таблице. После этого с учетом обратной связи участники начинают дорабатывать уже существующий алгоритм или создают совершенно новый.
В этом случае команды не получали никакой обратной связи. Они могли делать неограниченное количество попыток, но всякий раз у них не было никакой информации об их успешности, а также о положении дел у их конкурентов.
«Получается игра вслепую, чтобы к этому приспособиться, есть несколько вариантов, — рассказывает Артем Иванов. ― Один из классических путей — кросс-валидация, когда мы откладываем из размеченных данных какую-то часть, не обучаем на ней алгоритм, а только проверяем его. Проблема также в том, что любая работа с ДНК — это очень большие данные: на создание алгоритма, его обучение, проверку уходило около недели. За все время мы сделали около трех десятков попыток, но послали только лучшие».
Еще одним способом нащупать правильный путь стал поиск дополнительных данных. Команде ИТМО удалось найти в интернете еще одну подборку размеченных данных, чтобы также использовать ее для обучения и проверки своего анализатора.
«Самым важным в машинном обучении являются как раз данные, — объясняет Владимир Ульянцев. — Мы можем бесконечно тренировать алгоритм, но он не будет ничего стоить по сравнению с гигантами отрасли, обучающими на большом количестве данных. Поэтому мы принялись искать и загружать дополнительные данные в открытых источниках, которых не дали нам организаторы, но которые подходили бы нам. Возможно, за счет этого мы и выиграли».
Только предсказания мало
Помимо соревновательной, MEDIC имел также и научную составляющую ― даже более важную как для организаторов, так и для участников. По итогам турнира команды-победители приглашаются к сотрудничеству и созданию совместных публикаций в рецензируемых научных журналах. Поскольку представители Университета ИТМО победили в обеих номинациях, то они планируют включиться в эту работу.
«Задача диагностирования этих заболеваний очень острая, — подчеркивает Артем Иванов. — Пока в ходе соревнований мы просто выделили метагеномные признаки, по которым можно диагностировать заболевание. Было бы интересно понять, как и почему они связаны с развитием болезни. Как с любым медицинским исследованием: машинное обучение — это хорошо, но если мы дальше идем к какому-то врачу, нужно объяснить, а что у нашего черного ящика внутри ― он не может слепо верить какой-то модели».

Тяжело, но интересно
Как признается Артем Иванов, для него соревнования стали «большим и интересным опытом».
«Раньше с таким объемом данных работать не приходилось, это тяжело и интересно: надо понять, как данные запустить в алгоритм, как обрабатывать, надо следить за временем, чтобы не отставать от графика», ― говорит он.
Как считает Владимир Ульянцев, проведение таких соревнований имеет большое значение для сообщества тех, кто занимается изучением и лечением заболеваний на основе метагеномного анализа.
«На моей практике разбираться с диагнозами ― сложное дело. Диагноз — это зашумленные данные: один врач ставит один диагноз, другой врач — другой. Здорово, что такие соревнования начали проводить, и здорово, что мы его выиграли», — подытожил он.